在分子生物学和生物信息学领域,DNAman作为一款功能强大的序列分析软件,广泛应用于DNA和蛋白质序列的比对、进化分析以及结构预测等多个方面。本文将详细介绍如何使用DNAman进行二级结构预测以及如何进行进化树分析,帮助研究人员高效地进行基因和蛋白质的研究工作。
一、DNAman简介
DNAman是一款集成了多种序列分析工具的软件,支持多种生物分子的序列操作和分析。其主要功能包括序列比对、序列编辑、结构预测、进化树构建等。DNAman的用户界面友好,操作简便,适合生物学研究人员和生物信息学初学者使用。
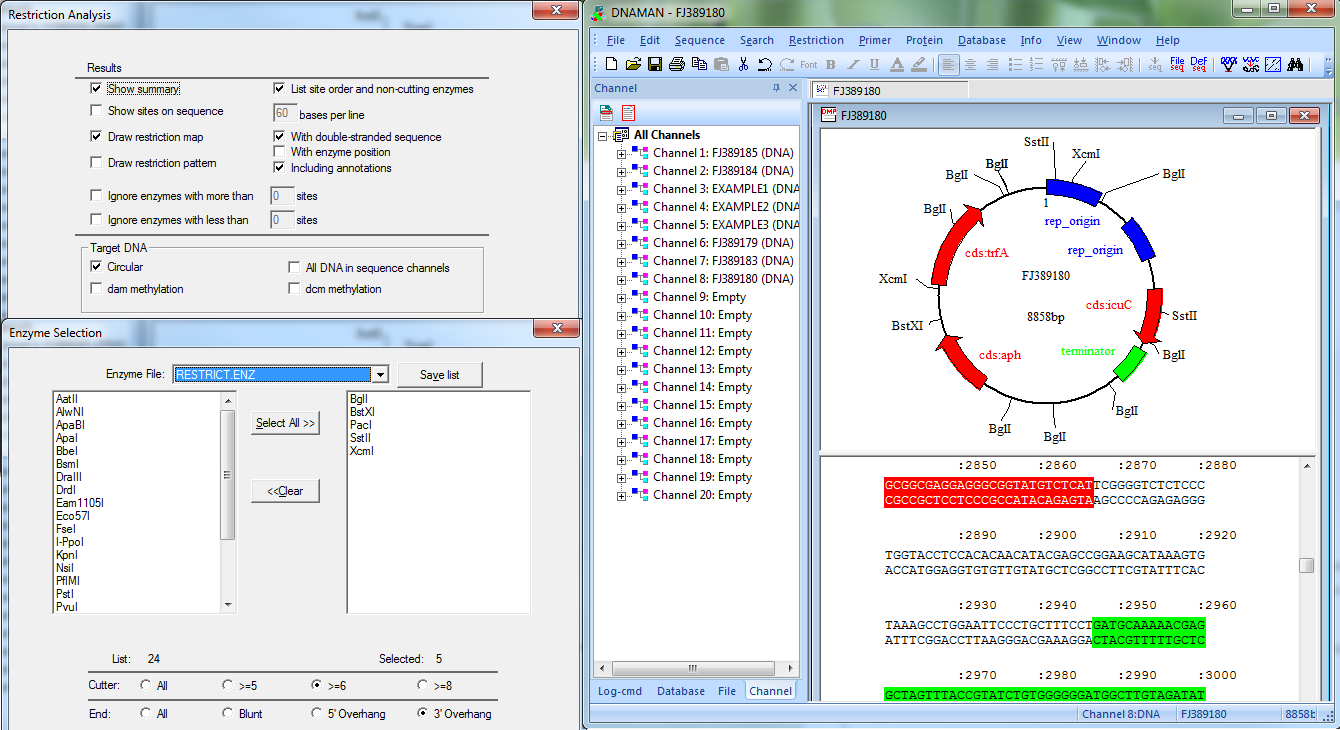
二、DNAman进行二级结构预测
二级结构预测是指根据一条生物分子的一级序列,预测其在空间中的局部结构,如DNA的发夹结构或蛋白质的α螺旋和β折叠等。使用DNAman进行二级结构预测的步骤如下:
1. 启动DNAman并导入序列
打开软件:启动DNAman软件。
导入序列:点击“文件”菜单,选择“打开”或“导入”功能,将需要预测二级结构的DNA或RNA序列导入软件中。支持的格式包括FASTA、GenBank等。
2. 选择二级结构预测工具
导航至预测模块:在主界面上,找到“分析”或“结构预测”选项卡。
选择预测类型:根据研究需求,选择相应的二级结构预测工具。例如,选择“DNA/RNA二级结构预测”功能。
3. 设置预测参数
选择算法:DNAman可能集成了多种预测算法,如最小自由能法(MFE)等。选择适合的算法以提高预测准确性。
调整参数:根据具体需求,调整预测参数,如温度、离子浓度等,这些参数会影响二级结构的稳定性和形成。
4. 运行预测并查看结果
启动预测:点击“开始预测”按钮,软件将根据设置的参数进行二级结构预测。
查看结果:预测完成后,结果将以图形化的形式展示在界面上。用户可以通过折叠线图或三维模型查看预测的二级结构。
5. 保存和导出结果
保存结果:点击“文件”菜单,选择“保存”或“另存为”,将预测结果保存到本地磁盘。
导出图像:如果需要在论文或报告中使用预测图,可以选择“导出图像”功能,将结构图导出为PNG、JPEG等格式。
6. 验证和优化
实验验证:二级结构预测结果应结合实验数据进行验证,如使用核磁共振(NMR)或X射线晶体学方法。
优化预测:根据实验结果,调整预测参数或选择其他预测算法,以提高预测的准确性和可靠性。
三、DNAman进行进化树分析教程
进化树分析是研究生物物种之间进化关系的重要工具。通过构建进化树,研究人员可以了解物种的进化历史和亲缘关系。以下是使用DNAman进行进化树分析的详细步骤:
1. 准备序列数据
收集序列:收集需要进行进化分析的DNA或蛋白质序列。确保序列的质量和完整性,以获得准确的分析结果。
格式整理:将收集到的序列整理为统一格式,如FASTA格式,方便导入DNAman进行分析。
2. 导入序列并进行多序列比对
导入序列:在DNAman主界面,点击“文件”菜单,选择“导入”功能,将准备好的序列文件导入软件中。
执行多序列比对:选择“比对”或“多序列比对”功能,使用ClustalW、MUSCLE等内置比对工具,对导入的序列进行比对。多序列比对是构建准确进化树的基础步骤。
3. 构建进化树
选择进化树构建方法:在比对完成后,选择“进化树构建”功能。DNAman通常支持多种进化树构建方法,如邻接法(Neighbor-Joining)、最大似然法(Maximum Likelihood)等。
设置构建参数:根据研究需求,设置进化树构建的参数,如替代模型、引导法(Bootstrap)次数等。这些参数会影响进化树的构建精度和可靠性。
运行构建程序:点击“构建”按钮,软件将根据设置的参数生成进化树。
4. 分析和可视化进化树
查看进化树:构建完成后,进化树将以图形化形式展示在界面上。用户可以通过缩放、旋转等操作,详细查看进化树的分支和节点。
注释与标记:根据需要,添加物种名称、节点支持值(如引导值)等注释信息,提升进化树的可读性和信息量。
保存和导出:将进化树保存为项目文件,或导出为图片格式(如PNG、SVG),方便在论文或报告中使用。
5. 解释和应用
解释进化关系:根据构建的进化树,分析物种之间的亲缘关系和进化路径,揭示物种的进化历史。
应用研究:将进化树分析结果应用于生态学研究、基因功能预测、物种保护等多个领域,促进科学研究的深入发展。
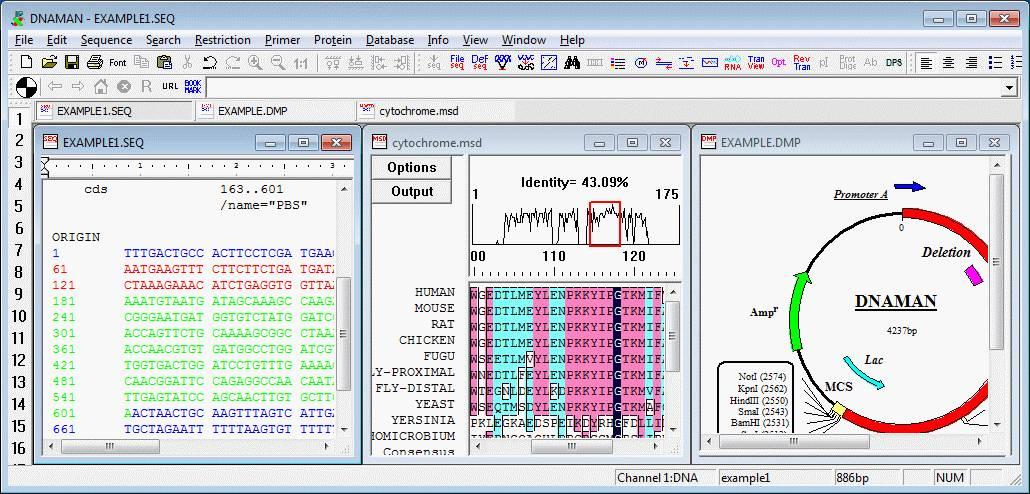
四、提高分析准确性的技巧
为了确保二级结构预测和进化树分析的准确性,以下是一些实用的技巧:
1. 数据质量控制
序列质量:确保导入的软件序列无误,避免含有错误或缺失的碱基或氨基酸。
去除冗余:在进行多序列比对前,去除冗余序列,减少分析复杂性,提高进化树构建的准确性。
2. 参数优化
适当选择算法:根据数据类型和研究需求,选择最适合的预测算法和进化树构建方法。
调整参数设置:根据实际情况,调整软件的参数设置,如比对窗口大小、进化模型等,优化分析结果。
3. 多方法验证
多次预测:对于二级结构预测,可以使用多种算法进行预测,综合分析结果,提高预测的可靠性。
多重验证:通过实验数据或其他软件工具验证进化树的构建结果,确保进化关系的准确性。
4. 学习和培训
熟悉软件功能:深入了解DNAman的软件功能和使用方法,通过官方文档和教程提升操作技能。
参加培训课程:参加相关的培训课程或工作坊,学习先进的序列分析和进化树构建技术,提升研究能力。
五、总结
DNAman作为一款功能强大的序列分析软件,在二级结构预测和进化树分析中发挥着重要作用。通过合理设置和优化分析参数,研究人员可以高效地进行基因和蛋白质的结构预测与进化关系研究。本文详细介绍了使用DNAman进行二级结构预测和进化树分析的步骤与技巧,旨在帮助用户充分利用软件功能,提升研究工作的效率和准确性。掌握这些方法和技巧,将为分子生物学和生物信息学研究提供有力支持,推动科学研究的不断进步。

